AB 15_2.3
Thema: Proteine – heute, gestern und morgen
zurück zur Übersicht: Arbeitsblätter (AB) Kapitel 2
Es ist richtig zu sagen, dass Leben nur durch die Aktivität von Proteinen möglich ist, da sie an fast jedem Vorgang in unserem Körper beteiligt sind. Ihre Bauanleitung wird von Tausenden protein-codierenden Genen gespeichert, die bei Bedarf aktiv werden.
Nicht zuletzt deshalb spielen Proteine in unserer heutigen Ernährung eine besondere Rolle. Diese ist im Kapitel 2.3 und in den Arbeitsmaterialien AB 1_2.3 bis AB 14_2.3 auf vielfältige Art und Weise deutlich gemacht worden. Auch wenn es noch weitere Aspekte gibt, unter denen sich die heutige Bedeutung von Proteinen beschreiben lässt, soll damit das „Heute“ (fast) abgehandelt sein.
Hinweise:
– Um den Stellenwert der Proteine in Zusammenhang mit den hier angesprochenen Forschungs- und Arbeitsbereichen und den zugehörigen Anwendungsgebieten vollständig zu erläutern, wären sehr umfangreiche Ausführungen notwendig. Bei den hier gemachten Aussagen handelt es sich nur um vereinfachte und verkürzte Darstellungen. Ausführlichere Informationen finden sich u.a. unter den angegebenen Links.
– Zum besseren Verständnis der folgenden Ausführungen sind Kenntnisse über den Aufbau der Proteine (AB 1_2.3) und den Ablauf der Proteinbiosynthese (AB 2_2.3) sinnvoll.
Wie konnte die Natur diese Vielfalt von Proteinen und damit unterschiedlichste Lebensformen bzw. Arten erschaffen?
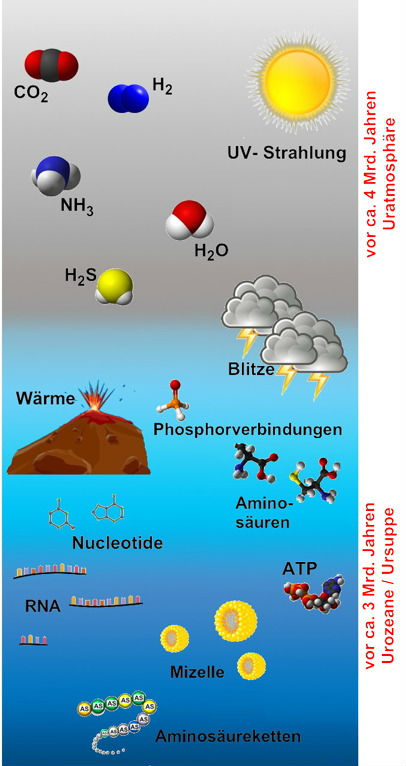
Zur Beantwortung dieser Frage muss man sich in Hinblick auf die Proteine in das „Gestern“ begeben, d.h. den Evolutionsprozess.
Im Folgenden werden einige kurze und vereinfachte Erläuterungen dazu gegeben. Dabei ist darauf hinzuweisen, dass die getroffenen Aussagen einerseits auf vielfältigen Untersuchungen und Experimenten beruhen, sie andererseits dennoch „nur“ im Sinne von Modellvorstellungen bzw. Theorien zu verstehen sind.
Im Erbmaterial (DNA) gespeicherte Informationen werden im Rahmen der Proteinbiosynthese (siehe AB 2_2.2 „Proteinbiosynthese und mehr“) in Proteine umgesetzt. Diese wiederum sind dafür verantwortlich, dass jeder Organismus bestimmte sichtbare bzw. messbare Merkmale aufweist (Formen, Größen, Farben, Stoffwechselprozesse).
Im Rahmen der Fortpflanzung werden die Informationen über die Proteine bzw. Merkmale über die DNA an die Nachkommen weitergegeben. Aufgrund verschiedener Vorgänge werden dabei die Informationen des Erbmaterials neu zusammengestellt bzw. geändert. Dementsprechend ändern sich in jeder Folgegeneration einer Art die auf der Grundlage der Proteinbiosynthese ausgebildeten Merkmale mehr oder weniger. Diese geänderten Merkmale mussten sich im Laufe der Evolution immer in der jeweiligen Umwelt bewähren. Taten sie das nicht, so wurden sie seltener an Nachkommen weitergegeben, passten sie gut in die Umwelt, so geschah dieses häufiger.
In diesem Zusammenspiel zwischen Erbmaterial, Fortpflanzung und Umwelt kam es zur Ausbildung immer neuer Merkmale in der sich über Jahrmillionen hinweg ständig ändernden Umwelt und damit zur Entstehung neuer Arten.
|
| Lösungen zu 1 u. 2:
zu 1. Alle drei Antworten sind richtig. Für Interessierte: zu 2.
|
Lernen vom “Gestern” für das “Morgen” – Proteine und unsere Zukunft
Zuvor wurde das Zusammenspiel von Erbmaterial (DNA) und Proteinen erwähnt. Heute besitzen Wissenschaftler die Fähigkeiten, beides gezielt zu verändern. Sie können somit aktiv in Stoffwechselprozesse eingreifen und gewissermaßen einen Evolutionsprozess nachahmen.
Der zugehörige Forschungs- und Arbeitsbereich nennt sich „Proteinengineering“ und bildet einen speziellen Teil der modernen Biotechnologie.
Im Rahmen der Arbeit an bzw. mit Proteinen werden auch Methoden der Gentechnologie eingesetzt. Schließlich sind das Genom bzw. Abschnitte der DNA zuständig für die Ausbildung bestimmter Proteine (siehe AB …).
Ziel der „Proteiningenieure“ ist, vorhandene Proteine an bestimmte Bedingungen besser anzupassen. Diese Arbeiten setzen Fähigkeiten zur exakten Untersuchung von Proteinstrukturen unter Verwendung chemischer und physikalischer Methoden voraus. Das Gelingen entsprechender Arbeiten ist außerdem nur durch den Einsatz leistungsstarker Computer und spezieller Computerprogramme, im Rahmen der sogenannten Bioinformatik, möglich.
Proteinengineering
Proteinengineering (Engineering = angewandte Ingenieurwissenschaft) ist ein Teilgebiet der modernen Biotechnologie und sehr eng mit der Gentechnologie verbunden. Entsprechende Tätigkeiten befassen sich mit der Analyse und Veränderung von in der Natur existierenden Proteinen oder der völligen Neukonstruktion bisher nicht vorhandener Proteine. Diese werden durch zufällige oder gezielte Eingriffe in die Aminosäuresequenz geschaffen. Nach erfolgreichen Tests werden entsprechende Produkte (z.B. Enzyme, Antikörper) hergestellt. Diese dienen nicht nur der Grundlagenforschung, sondern werden im Rahmen der Stoffwechselprozesse in Zellen bzw. Organismen zur Herstellung von Stoffen verschiedenster Art genutzt. Weitere Einsatzmöglichkeiten liegen in der Diagnose und Therapie von Krankheiten.
Langfristig wird angestrebt, auf diesen Wegen möglichst viele zelluläre Prozesse vollständig zu verstehen und in diese auch regulierend eingreifen zu können.
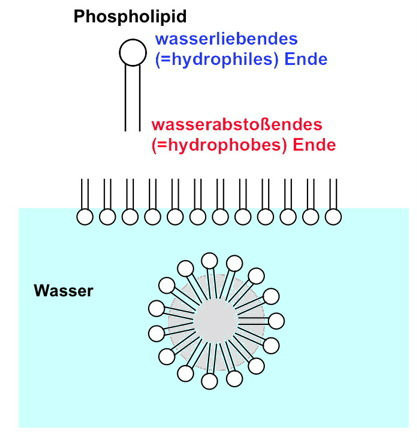
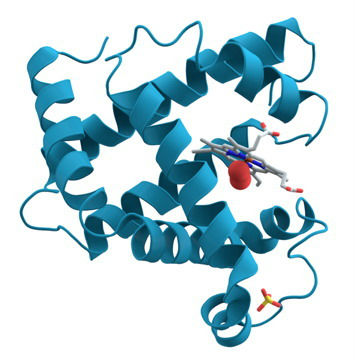
| Das klassische oder evolutionäre Proteinengineering ahmt die Vorgänge der Evolution nach. Zufällige Veränderungen in der DNA (=Mutationen) führen über eine entsprechende RNA zu einer neuen Aminosäuresequenz und damit zu einem neuen Protein. Dieses wird darauf getestet, ob es in Hinblick auf die gewünschte Wirkung zu verwenden ist. Dieser Vorgang wird sehr häufig durchgeführt, in der Hoffnung ein neues geeignetes Protein zu finden. Für diese Vorgehensweise sind keinerlei Kenntnisse der Strukturstufen eines Proteins (siehe …) notwendig. Diese Vorgehensweise ist sehr aufwändig bzw. langwierig und entspricht ein wenig dem Weg von Versuch und Irrtum.Ein anderer Weg ist das sogenannte Proteindesign. Dieses setzt genaue Kenntnisse über das zu verändernde Protein voraus (Abb. 15_2.3.-2). Dazu gehören nicht nur die Aminosäuresequenz, sondern auch die Faltungen und andere Besonderheiten des Proteins. Diese werden zunächst am Computer dreidimensional dargestellt. Die gewünschten Veränderungen werden digital erzeugt, bis die gewünschten neuen Strukturen vorliegen. Erst dann kommen biochemische und physikalische Methoden zum Einsatz, mit deren Hilfe das veränderte Protein hergestellt wird. Die Tests in Hinblick auf die Wirksamkeit des neuen Proteins sind nicht so aufwändig, wie bei der klassischen Vorgehensweise. Ganz neue Wege des Proteindesigns bestehen darin, dass ein Protein vollständig neu am Rechner entworfen wird oder dass sogar mit veränderten neuen Aminosäuren experimentiert wird. |
Auf der Grundlage der aktiven Gestaltung von Proteinen wurden und werden Produkte z.B. zur Krebs- und Malariabekämpfung, zur Thromboseverhinderung, zur Behandlung von Schlafstörungen entwickelt. Es wird davon ausgegangen, dass zukünftig mithilfe der zuvor genannten Verfahren vor allem eine Vielzahl von Diagnose- und Therapiemöglichkeiten für die verschiedensten Bedürfnisse bzw. Krankheiten geschaffen werden.
Nicht verwunderlich, dass der vermutete Marktwert – allein für Arzneimittel, die auf der Grundlage der genannten Wege produziert werden – für das Jahr 2017 mit 1,6 Mrd. $ und für 2025 mit mehr als 3 Mrd. $ angegeben wird.
Die Bekämpfung von bakteriellen Infektionen wird immer schwieriger, da immer mehr Bakterien eine Antibiotikaresistenz (= Widerstandskraft gegen die Wirkung eines Antibiotikums) aufweisen. Forschern ist aufgefallen, dass der Speichel von Komodowaranen auf den Marshall-Inseln einerseits sehr viele Bakterien enthält, die Entzündungen auslösen. Andererseits fiel auf, dass bei Kämpfen zwischen den Waranen auch größere Wunden keine Entzündungsherde zeigten, obwohl diese durch die Bakterien aus dem Speichel der gegnerischen Warane infiziert gewesen sein mussten. |
||
| Zecken können sich auf jedem Untergrund festhalten. Bisher war bekannt, dass sie dazu spitze Krallen und dazwischenliegende Haftpolster benutzen. Nun haben Forscher der Universitäten Dresden und Kiel festgestellt, dass sowohl die Krallen als auch die Haftpolster mit dem elastischen Protein „Resilin“ gefüllt bzw. von diesem umgeben sind. Dieses wirkt wie ein Haftpad und entwickelt ein Haltevermögen, das etwa dem 500fachen Körpergewicht der Zecke entspricht. Vor allem in der operativen Medizin sucht man nach Stoffen, die einerseits sehr elastisch sind, andererseits extrem viel aushalten und nicht zerbrechen. Benötigt werden solche Stoffe z.B. in Zusammenhang mit Hüft- oder Kniescheibenersatz oder Gefäßprothesen (= Ader-Ersatz).siehe auch: https://tu-dresden.de/wie-zecken-halt-finden |
| Lösungen zu 3.:
Wie kann man die im Waranblut gefundenen antimikrobiellen Peptide auch für den Menschen im Rahmen der Bekämpfung von bakteriell bedingten Entzündungen nutzen? Auf der Grundlage des bei der Zecke gefundenen Resilins versuchen Proteindesigner ein entsprechendes Protein zu entwickeln, das einerseits die Eigenschaften des ursprünglichen Proteins besitzt. Andererseits muss es so an den menschlichen Organismus angepasst werden, dass es nicht als Fremdprotein abgestoßen wird. |
Nicht nur in medizinisch-pharmazeutischen Bereichen wurde und wird erfolgreich und intensiv mit Proteinen gearbeitet. Seit einigen Jahren werden entsprechende Kenntnisse zunehmend genutzt, um gezielt Mikroorganismen (Bakterien, Pilz, Algen) – genau genommen deren Stoffwechselwege – zu verändern und an die Bedürfnisse der Menschen aktiv anzupassen.
Immer häufiger werden in Zusammenhängen mit den zuvor genannten Aktivitäten die Begriffe „Metabolic Engineering“ (Syn.: „Pathawaydesign“) genannt. Diese beziehen sich hauptsächlich auf Mikroorganismen und Pflanzen, aber auch auf Zellkulturen anderer Organismen.
Der folgende Link macht die Vielfalt der möglichen Nutzung von mehr oder weniger angepassten Mikroorganismen deutlich.
| https://biooekonomie.de/themen/dossiers/mikrobielle-zellfabriken-die-spitzenkraefte-der-biotechnologie(Zugriff: 2021-09-15) |
Die vorgenommenen Veränderungen betreffen in erster Linie Enzyme, deren
|
Anwendungsbereiche des Metabolic Engineering finden sich z.B. in der Lebensmittelproduktion und der Erzeugung von Biokraftstoffen (siehe AB EB_6 “Projekt: Kohlenstoffdioxid-Kreislauf, Biokraftstoffe und Enzyme (wird z.Zt. erstellt)) – eigentlich zunehmend überall dort, wo mit Hilfe von Mikroorganismen (Bakterien, Hefen, Algen) Produkte hergestellt werden.
Der Kern vieler zuvor genannter Aktivitäten besteht letztlich darin, „benutzerdefinierte“ Vorgaben bzw. Ziele durch vorzunehmende Änderungen im Genom zu verwirklichen. Dieses wird vor allem seit der Beherrschung der CRISPR-Methode verstärkt möglich.
Die CRISPR/Cas-Methode ist eine molekularbiologische Methode, um DNA gezielt zu schneiden und zu verändern. Gene können dadurch eingefügt, entfernt oder ausgeschaltet werden. Auch Nucleotide in einem Gen können geändert werden. Aufgrund der einfachen Durchführung und der geringen Kosten wird die CRISPR/Cas-Methode zunehmend in der Forschung und Entwicklung eingesetzt (siehe auch Link unten).
Ein weiterer Begriff, der alles zuvor Genannte einschließt und noch darüber hinausgeht lautet „Synthetische Biologie“. Dieser Arbeitsbereich betrifft ebenfalls schwerpunktmäßig die Ebene der Mikroorganismen und Pflanzen.
Synthetische Biologie
Mit Hilfe bekannter Methoden sollen neue künstliche biologische Systeme geschaffen werden und in diesen Zusammenhängen
– vorhandene Stoffwechselprozesse nicht nur verändert, sondern im Sinne kontrollierbarer Schaltkreise (= biochemische Signalwege) konstruiert werden, um so
– eine völlige Neuentwicklung ganzer Organismen unter Verwendung von molekularen Modulen zu ermöglichen.
Das Ziel ist, Werkzeuge zu schaffen, mit deren Hilfe man chemische Grundstoffe und Proteine gezielt herstellen kann.
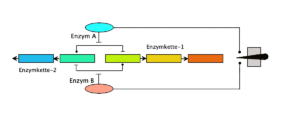
Abb. AB 15_2.3-6 Animation: Denkmodell einer Schaltkreis-Steuerung der Stoffwechselaktivität (vereinfacht)
Hinweis: Die zweite Enzymkette und die damit verbundene Stoffproduktion sind nur angedeutet.
| Zum Starten der Animation anklicken! | |
|
|
| Erklärung zu 5. (vereinfacht):
Eigentlich geht es – sehr vereinfacht betrachtet – nur darum, ob über zwei verschiedene Leitung die blaue Lampe oder die rote Lampe angeschaltet wird. |
Teile der Wissenschaft verbinden mit der synthetischen Biologie noch wesentlich weitergehende Aktivitäten. Im Idealfall geht es darum, einen sogenannten „Minimalorganismus“ zu erzeugen. Dieser Organismus soll nur die wichtigsten Gene enthalten, die ihn am Leben erhalten. Einzelne konstruierte Stoffwechselwege sollen dann – je nach Zielsetzung – das Genom des Minimalorganismus ergänzen, um Stoffe zu produzieren.
Der radikalste Denkansatz der synthetischen Biologie ist die sogenannte „Xenobiologie“(griech.: xénos / „Fremder, Gast“). Dabei ist die Konstruktion von künstlichen Mikroorganismen vorgesehen, die einen derartig verändertes Genom und/oder nicht natürlich vorkommende Aminosäuren bzw. Proteine aufweisen. Auf diesem Weg hoffen Wissenschaftler, vollkommen neuartige Stoffe, z.B. Wirkstoffe gegen resistente Krankenhauskeime, zu produzieren.
Alles zuvor Gesagte macht die Bedeutung der Proteine – insbesondere der Enzyme – sehr deutlich. Sie werden maßgeblich an der Entwicklung verschiedenster zukünftiger Lebensgrundlagen beteiligt sein.
Dabei darf aber nicht übersehen werden, dass die genannten Forschungs- und Anwendungsbereiche unter ökologischen und moralisch-ethischen Gesichtspunkten kritisch diskutiert werden müssen. Exemplarisch wird dieses am Beispiel manipulierter Mikroorganismen für die Biokraftstoff-Herstellung erläutert. (siehe AB EB_6 “Projekt: Kohlenstoffdioxid-Kreislauf, Biokraftstoffe und Enzyme / (wird z.Zt. erstellt)).
Aufgrund zunehmender Komplexität von Forschung und Entwicklung gewinnt ein weiterer Begriff in Theorie und Praxis zunehmend an Bedeutung, die Systembiologie. Sie wird inzwischen auch als eigene Wissenschaftsdisziplin angesehen und gelehrt.
Systembiologie (engl.: integrative biology)
Die Systembiologie ist ein ganzheitlicher Ansatz, der versucht Prozesse auf unterschiedlichen Ebenen (z.B. Molekül, chemische Prozesse, Zellorganelle, Zelle, Gewebe, Organismus, …) unter Beteiligung verschiedenster wissenschaftlicher Disziplinen zu analysieren, zu verändern, zu schaffen, zu modellieren, zu simulieren und zu steuern.
Die darauf basierende Forschung und Entwicklung vermeiden die Zersplitterung von Wissen und damit letztlich auch überflüssige Kosten. Am häufigsten sind Schnittmengen zwischen den Naturwissenschaften, den Ingenieurswissenschaften und der Informatik. Aber auch die Geisteswissenschaften, wie Psychologie und Soziologie werden z.B. im Kontext gesellschaftlicher Akzeptanz oder Folgen einbezogen. In den unterschiedlichsten Zusammenhängen nimmt die Ökonomie Einfluss auf systembiologisches Arbeiten.
Anwendungsbereiche sind neben der Grundlagenforschung vor allem die Medizin, die Landwirtschaft und verschiedenste Industriezweige.
Die folgende Linkliste ermöglicht die genauere Beschäftigung mit den zuvor behandelten Themen.

Dieses Werk ist lizenziert unter einer Creative Commons Namensnennung – Weitergabe unter gleichen Bedingungen 4.0 International Lizenz.